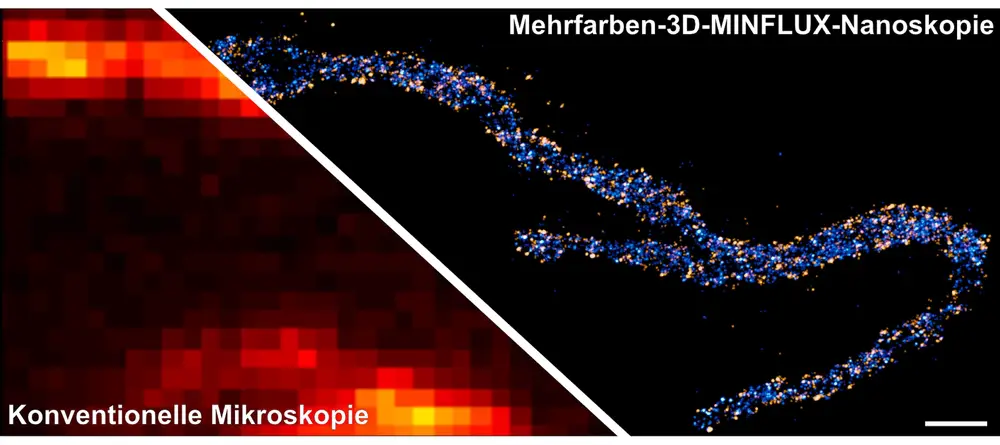
Die von Nobelpreisträger Stefan Hell und seinem Team entwickelte MINFLUX-Nanoskopie ermöglicht, fluoreszierende Moleküle mit Licht getrennt abzubilden, die nur ein paar Nanometer (millionstel Millimeter) voneinander entfernt sind. Diese Technik ist damit hundertmal schärfer als die herkömmliche Fluoreszenz-Lichtmikroskopie. Forscherinnen und Forscher um Stefan Hell und Stefan Jakobs haben mit dieser Methode erstmals die Molekülverteilung innerhalb einzelner Proteinkomplexe eines Zellorganells sichtbar gemacht – und das mehrfarbig und in 3D. Damit eröffnet die MINFLUX-Nanoskopie neue Anwendungsmöglichkeiten in der Biologie und Medizin.
Für die MINFLUX-Nanoskopie hatte Stefan Hell die Stärken der beiden bis dato hochauflösendsten Fluoreszenz-Nanoskopie-Techniken auf originelle Weise zusammengeführt: PALM/STORM und das von ihm entwickelte STED-Verfahren, für das der Physiker 2014 den Nobelpreis für Chemie erhielt. Die 2016 vorgestellte MINFLUX-Methode erreicht erstmals eine Trennschärfe von wenigen Nanometern und verfolgt Biomoleküle, die sich in der Zelle bewegen, bis zu hundertmal schneller als herkömmliche Geräte. Die Technik hat sein Team seitdem für die biologische Anwendung optimiert.
„In den letzten Jahren haben meine Kollegen Klaus Gwosch, Francisco Balzarotti und ich daran gearbeitet, fluoreszierende Moleküle in Zellen mit maximaler, molekularer Auflösung in zwei Farben und 3D mithilfe von MINFLUX sichtbar zu machen. Das ist uns kürzlich gelungen“, erzählt Jasmin Pape, wissenschaftliche Mitarbeiterin in der Abteilung NanoBiophotonik von Stefan Hell. „Da Biologen häufig komplexere Proben analysieren, haben wir die Methode auch für diese Anforderung angepasst. Wir können jetzt selbst dicht gepackte Strukturen in Organellen von Zellen sichtbar machen und quantitativ untersuchen.“
Schlüsseltechnologie in der Zellbiologie
MINFLUX hat den Praxistest erfolgreich gemeistert. „Mit dieser herausragenden Technik haben wir in den Kraftwerken der Zelle, den Mitochondrien, eine Auflösung von nur wenigen Nanometern erreicht“, berichtet Jakobs, Forschungsgruppenleiter am Max-Planck-Institut (MPI) für biophysikalische Chemie und Professor an der Klinik für Neurologie der Universitätsmedizin Göttingen (UMG). „Dies ist die höchste Auflösung an komplexen intrazellulären Strukturen, die in 3D je erreicht wurde. Ich freue mich sehr über die erste echte Anwendung von MINFLUX“, so Hell, Direktor am MPI für biophysikalische Chemie und am MPI für medizinische Forschung. „Die hervorragende Kooperation an unserem Institut, in dem Biologie, Chemie und Physik unter einem Dach vereint sind, hat diesen Erfolg ermöglicht. Ich bin überzeugt, dass MINFLUX eine Schlüsseltechnologie für die Zellbiologie in den nächsten Jahren sein wird, denn damit kann man Zellen molekular kartografieren.“
Die Auflösung der MINFLUX-Nanoskopie ist so gut, dass die Wissenschaftlerinnen und Wissenschaftler jeden Fehler bei der Probenvorbereitung unter dem Mikroskop sofort sehen. „Damit werden die Probenvorbereitung und das Markieren zum limitierenden Faktor – und nicht mehr wie bisher die Auflösung der Mikroskopie“, erklärt Till Stephan, wissenschaftlicher Mitarbeiter von Jakobs. „Entscheidend für eine gute Mikroskopie-Aufnahme ist, wie gut wir die Struktur, die wir untersuchen wollen, während der Probenvorbereitung erhalten können und wie nahe wir den Farbstoffmarker an die Zielproteine anheften können.“
Neue molekulare Details aus Mitochondrien
Jakobs erforscht mit seinen Mitarbeiterinnen und Mitarbeitern seit vielen Jahren die Struktur und Funktion von Mitochondrien. Die molekularen Kraftwerke liefern die nötige Energie, um den Stoffwechsel unserer Körperzellen in Gang zu halten. Dazu besitzen sie einen einzigartigen Aufbau aus einer glatten äußeren und einer fingerförmig eingefalteten inneren Membran. Die Proteinmaschinerie, die im menschlichen Körper pro Tag etwa 75 Kilogramm des Energiespeichermoleküls ATP liefert, sitzt in den inneren Einstülpungen, Cristae genannt. Wo Cristae auf die innere Grenzmembran treffen – an den sogenannten Crista Junctions – ist eine weitere wichtige molekulare Maschinerie namens MICOS am Werk. MICOS besteht aus mehreren Proteinen, die im Team die innere Membran der Mitochondrien in die richtige Form biegen.
Unter dem MINFLUX-Nanoskop entdeckte das Team um Jakobs jetzt überraschende Details der Crista Junctions. „Unsere Aufnahmen legen nahe, dass sich die MICOS-Proteine um die Crista Junctions äußerst heterogen verteilen“, berichtet der Biologe. Seine Gruppe wird mithilfe der MINFLUX-Nanoskopie untersuchen, wie das Zusammenspiel der MICOS-Schlüsselproteine gesteuert wird. Da viele Erkrankungen des Nervensystems und des Muskelapparats mit einer gestörten Mitochondrienarchitektur einhergehen, können neue Erkenntnisse dazu beitragen, besser zu verstehen, wie derartige Krankheiten entstehen.
Kommerzielles MINFLUX-Nanoskop
Für seine weitere Forschung wird das Team um Jakobs zukünftig auch ein kommerzielles MINFLUX-Nanoskop einsetzen: Eines von nur vier Geräten weltweit wird in den nächsten Wochen in Göttingen aufgestellt. Die Deutsche Forschungsgemeinschaft fördert das von Jakobs beantragte Projekt mit 2,3 Millionen Euro. Das neue Hochleistungs-Nanoskop wird Teil der Ausstattung im Labor des Biologen an der Universitätsmedizin Göttingen sein, auch Forscherkolleginnen und -kollegen am Göttingen Campus können es nutzen. „Die Bandbreite der Anwendung der MINFLUX-Nanoskopie reicht von der biomolekularen Chemie über die Hörforschung bis hin zur Kardiologie und Neurologie und wird den Forschungsstandort Göttingen weiter stärken“, sagt Jakobs.
Die Animation einer MINFLUX-Nanoskopie-Aufnahme zeigt ein Mitochondrium aus einer Hautzelle. Eine Untereinheit des MICOS-Komplexes ist in orange angefärbt, eine Untereinheit der ATP-Synthase in blau. | Animation: Till Stephan & Jasmin Pape / Max-Planck-institut für biophysikalische Chemie
Originalpublikation:
Jasmin K. Pape, Till Stephan, Francisco Balzarotti, Rebecca Büchner, Felix Lange, Dietmar Riedel, Stefan Jakobs, Stefan W. Hell: Multicolor 3D MINFLUX nanoscopy of mitochondrial MICOS proteins. Proceedings of the National Academy of Sciences USA, 11. August 2020,
https://doi.org/10.1073/pnas.2009364117