Mehr als ein Viertel aller Proteine einer Zelle finden sich in den Membranen. Um dort ihre Aufgabe erfüllen zu können, müssen Membranproteine von ihrer Produktionsstätte sicher zu ihrem Bestimmungsort transportiert und korrekt in die Zielmembran eingebaut werden. Wissenschaftlern der Universität Heidelberg ist es gelungen, die dreidimensionale Struktur einer molekularen Maschine zu entschlüsseln, die für den korrekten Einbau von sogenannten „tail-anchored“-Membranproteinen zuständig ist.
Mehr als ein Viertel aller Proteine einer Zelle finden sich in den Membranen. Dort haben sie lebenswichtige Funktionen. Um ihre Aufgabe zu erfüllen, müssen Membranproteine von ihrer Produktionsstätte in der Zelle sicher zu ihrem Bestimmungsort transportiert und korrekt in die Zielmembran eingebaut werden. Wissenschaftlern des Biochemie-Zentrums der Universität Heidelberg (BZH) ist es gelungen, die dreidimensionale Struktur einer molekularen Maschine zu entschlüsseln, die für den korrekten Einbau einer wichtigen Familie von Membranproteinen – die sogenannten „tail-anchored“-Membranproteine oder kurz TA-Proteine – zuständig ist.
Ein erwachsener Mensch besteht aus schätzungsweise 100 Billionen Zellen. Jede einzelne davon enthält unzählige Proteine, die als Konstrukteure und Akteure des Lebens vielfältige Aufgaben wahrnehmen. Ein großer Teil der Proteine einer Zelle sind Membranproteine, also Bestandteile der feinen Häutchen (lateinisch membrana), die jede Zelle, aber auch alle kleinen Organe – die Organellen – in ihrem Innern umhüllen. Membranproteine formen beispielsweise Kanäle oder Poren und erfüllen grundlegende Arbeiten wie den Stofftransport oder die Signalübermittlung. Vom korrekten Einbau, der Insertion, hängt ab, ob das Membranprotein seine biologischen Aufgaben erfüllen kann. Davon ist auch die ordnungsgemäße Funktion der Zelle abhängig. Doch wie wird gewährleistet, dass das Protein an die richtige Membran gelangt und dort an der richtigen Stelle in die Membran integriert wird?
Bedeutende Membranprotein-Familie
Für den zielgerichteten Transport und die korrekte Insertion sind bestimmte Signaturen wichtig – kleine Abschnitte der Proteine, die gleichsam als Postleitzahlen fungieren. Sie werden von molekularen Sortiermaschinen erkannt, die das Protein seinem Bestimmungsort zuweisen. Bei manchen Proteinen findet sich die Signatur am Ende des Moleküls: Die Wissenschaftler sprechen englisch von „tail-anchored“-Membranproteinen, kurz TA-Proteinen. Diese bedeutende Membranprotein-Familie ist an zahlreichen zellulären Prozessen beteiligt, etwa der Fusion von Membranen oder der Apoptose, dem programmierten Zelltod.
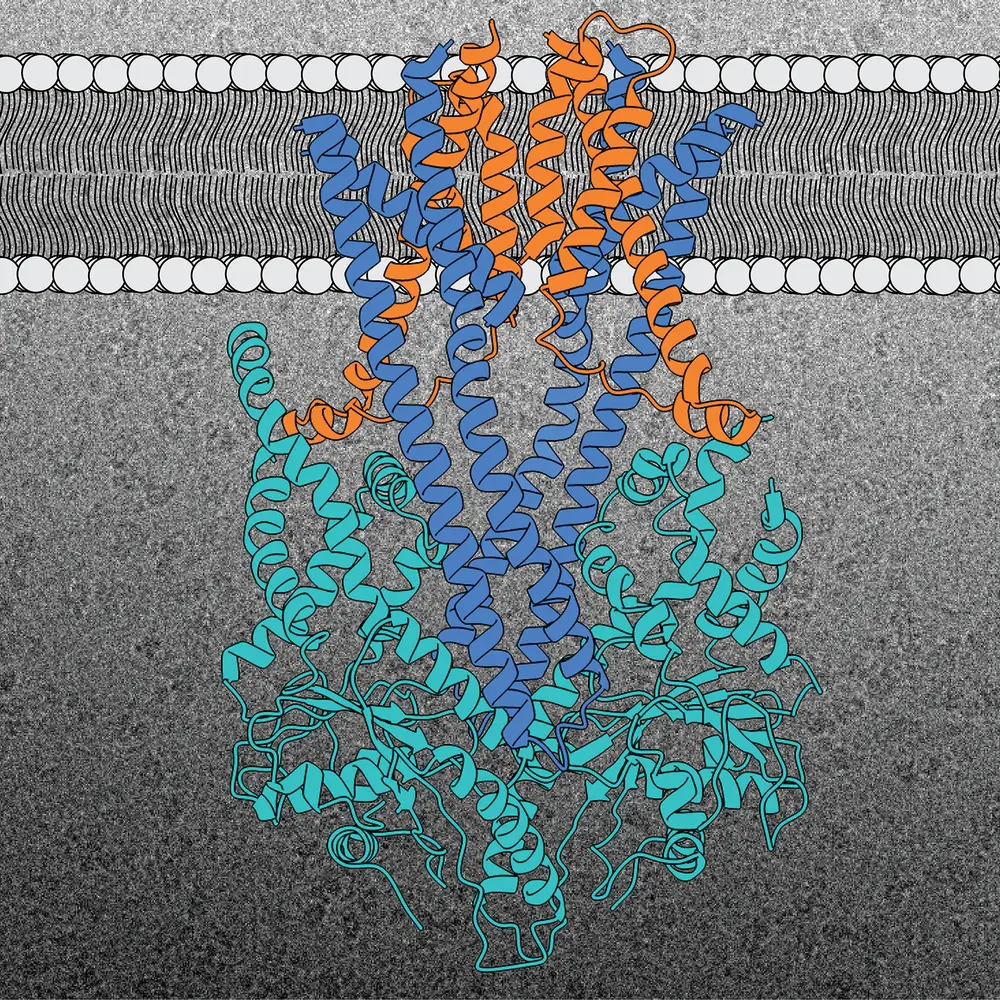
Wissenschaftler des BZH unter der Leitung von Prof. Dr. Irmgard Sinning konnten unlängst die dreidimensionale Struktur derjenigen molekularen Maschine entschlüsseln, die TA-Proteine in die Membranen des Endoplasmatischen Retikulum (ER) einbaut – ein wichtiges Verteilungsnetz im Innern der Zelle, das mit allen anderen Organellen verbunden ist. Für ihre Strukturanalysen nutzten die BZH-Forscher die Kryo-Elektronenmikroskopie (Kryo-EM), eine Methode, die 2017 mit dem Nobelpreis für Chemie ausgezeichnet wurde. „Derart hochaufgelöste Strukturinformationen sind von entscheidender Bedeutung, um die finalen Schritte bei der Insertion der Proteine in die ER-Membran zu verstehen“, erklärt Prof. Sinning, die eine Forschungsgruppe am BZH leitet.
GET-Insertionsmaschine im Laufe der Evolution kaum verändert
Verantwortlich für das korrekte Einfügen der TA-Proteine in die Membran des Endoplasmatischen Retikulums ist die sogenannte GET-Insertionsmaschine. Dabei steht die Abkürzung GET für „Guided Entry of Tail-anchored membrane proteins“. Diese Insertionsmaschine hat sich im Laufe der Evolution von der Hefe bis zum Menschen kaum verändert, und besteht aus drei Proteinbausteinen: Zwei sitzen in der ER-Membran und formen dort eine Art Tasche (Get1 und Get2). Der dritte Proteinbaustein (Get3) befindet sich außerhalb der Membran und fungiert als Zulieferer. Alle drei Bauteile der GET-Insertionsmaschine sind entscheidend für den korrekten Einbau des TA-Proteins in die Zielmembran: Get2 übernimmt das Protein vom Zulieferer und schiebt es quasi über die Tasche in die Membran. Dieses unerwartete Detail der Interaktion von Get2 und Get3 haben die Heidelberger Wissenschaftler durch ihre Proteinstrukturanalysen entdeckt. Weitere Analysen zeigten, dass stets zwei Kopien der Insertionsmaschine eng zusammenarbeiten, was die Effizienz der Integration des Proteins in die Membran erhöht. „Die GET-Insertionsmaschine ermöglicht den TA-Proteinen einen energetisch günstigen Zugang zur Membran“, so Prof. Sinning.
„Derart kleine Membranproteine, wie wir sie in der GET-Insertionsmaschine finden, sind eine Herausforderung für die Strukturbiologie. Für unsere Forschungsarbeiten waren daher innovative Ideen notwendig“, betont die Strukturbiologin Dr. Melanie McDowell. Erst seit einigen Jahren erlauben technische Verbesserungen in Kryo-EM, die Struktur von immer kleineren Proteinkomplexen immer detaillierter zu bestimmen. Die Universität Heidelberg hat daher ein „Kryo-EM-Netzwerk“ (HDcryoNet) etabliert, das es ermöglicht, auch die Struktur kleiner Membranprotein-Komplexe wie der GET-Insertionsmaschine zu analysieren. Prof. Sinning und Dr. McDowell sind überzeugt, dass ihre neuen Daten ein lange gesuchtes Puzzleteil sind, mit dem es gelingen kann, das Bild vom Proteintransport in der Zelle und der Insertion von Proteinen in die Membran zu vervollständigen.
Die Studie, an der auch Wissenschaftler der Universität Oxford (Großbritannien) und der Universitätsmedizin Göttingen beteiligt waren, wurde überwiegend von der Deutschen Forschungsgemeinschaft gefördert. Die Forschungsergebnisse wurden in der Fachzeitschrift „Molecular Cell“ veröffentlicht.
Originalpublikation:
M.A. McDowell, M. Heimes, F. Fiorentino, S. Mehmood, Á. Farkas, J. Coy-Vergara, D. Wu, V. Schmid, R. Heinze, J. R. Bolla, K. Wild, D. Flemming, S. Pfeffer, B. Schwappach, C.V. Robinson, & I. Sinning: „Structural basis of tail-anchored membrane protein biogenesis by the GET insertase complex“. Molecular Cell (2020) 80: 72-86.e7,
DOI: 10.1016/j.molcel.2020.08.012